本人受《人工智能导论》课程作业的启发,尝试使用遗传算法求解旅行商问题,并在本文将过程中遇到的问题和受到的启发记录下来。
后续会将本次实践的所有文件上传github。
遗传算法与旅行商问题
遗传算法
遗传算法(Generation Algorythm, GA)是一种随机全局搜索优化方法,其基本原理是模仿自然界中的进化过程,通过选择、交叉和变异等操作,从一个初始种群中产生更优的个体,从而寻找问题的最优解或近似最优解。
遗传算法的基本步骤如下:
- 将待解决问题的解抽象成DNA的形式;
- 初始化一个随机种群,种群中的个体均携带一条(或多条)DNA。于是,每个个体就代表了原问题的一个解;
- 根据个体的DNA来计算该个体的适应度,一般通过评估DNA所代表的解在原问题中的优劣来确定DNA的适应度;
- 对种群进行选择操作,即尽可能淘汰种群中适应度低的个体,使得携带代表原问题的优解的DNA的个体得以存活;
- 对种群进行交叉和变异操作,增加种群多样性,以提高代表更优的解的DNA出现的概率;
- 记录这一代的参数(比如适应度最高的个体的DNA)并评估;
- 重复3~6,直到满足预设的终止条件。
旅行商问题
旅行商问题(Traveling Salesman Problem, TSP),是一个经典的NP难题。问题描述为:给定一批城市以及每两个城市之间的距离,找到一条恰好经过每个城市一次(最后要回到起点)且路程最短的路线。在图论中,也可以这样描述:找到一个n阶带权完全图的权值总和最小的哈密顿回路。
为节省时间开销,一般只求解旅行商问题的近似最优解,本文就将提供一种用遗传算法求解旅行商问题的思路。
思路构建
1. 实现“解”到“DNA”的转化
首先要用抽象化的数学符号来表示原问题的对象:
- 将n个城市按0 ~ (n-1)编号,构造一个对角元素均为0的n阶对称方阵$M$,其元素$m_{ij}(i, j \in [0, n-1])$代表城市i到城市j的距离
- 用形如$C = \{c_0, c_1, \cdots , c_{n-1}\}$的序列代表一个解,表示$城市c_0 \rightarrow 城市c_1 \rightarrow \cdots \rightarrow 城市c_{n-1} \rightarrow 城市c_0$的一条路径
- 上述路径的路程即为$D(C) = \sum\limits_{i=0}^{n} m_{c_i c_{(i+1) \% n}} $
接下来将解$C = \{c_0, c_1, \cdots , c_{n-1}\}$转换成DNA的形式,一种常见的处理方式是,构造一个从解空间到一个二进制串空间的映射,但由于这个问题中解的一些性质(后续会解释),这个处理方式是不合适的,故本次实践中,保留了解的基本形式,用一个n维向量表示DNA。为了方便描述,下文用$C = \{c_0, c_1, \cdots , c_{n-1}\}$指代解和其对应的DNA,以及携带该DNA的个体,而基因则指代该序列中的单个元素$c_i$
2. 确定适应度函数和选择策略
- 适应度作为选择的唯一指标,确定一个能正确反映个体优劣的适应度函数是很重要的。显然,DNA所代表的路径路程越短的个体适应度越高,于是可以直接使用路程的倒数作为适应度。
- 也就是说,若DNA为 $C = \{c_0, c_1, \cdots , c_{n-1}\}$,适应度就等于:
- 得到每个个体的适应度后,就可以制定选择策略了。这里采用经典的轮盘赌选择(Roulette-wheel selection)策略,即个体的存活率(被选择率)等于个体适应度与种群总适应度的比值。
- 也就是说,若种群$P = \{C_0, C_1, \cdots, C_m \}$,个体的存活率等于
3. 确定交叉和变异策略
上文提到,一般会将解空间映射成一个二进制串空间,这样的DNA形式与自然界的DNA形式更为吻合,交叉与变异操作的实现也更简单与直观:交叉可以直接用DNA的子串进行操作,变异可以直接将DNA的某位取反。
但是在这个问题中,若采用上述的方案则无法保证解$C = \{c_0, c_1, \cdots , c_{n-1}\}$中元素的唯一性(这是为了保证能走遍所有城市),所以DNA就必须与解形式相同。
那么为了保证DNA经过交叉和变异操作后,其代表的解仍合法,我们需要制定特殊的交叉与变异策略。
使用部分映射交叉(Partial Mapping Crossover, PMX)方法实现交叉操作:首先从父DNA中随机选择一个连续的基因序列复制到子DNA相同位置,然后遍历母DNA,将子DNA中未具有的基因按顺序添加到子DNA中。
- 设父、母、子分别为$C_f, C_m, C_c$
- 随机确定复制范围(a, b),$c_{ci} = c_{fi}, i\in [a, b]$
- $c_{cj} = c_{mk}, j\in [0, a)\cup(b, n-1], k\in[0, n-1], c_{mk}\notin C_c$
变异则可以通过交换DNA任意两个基因的位置实现
代码实现
编写并运行
create_matrix.py脚本, 将表示城市间距离的矩阵保存成npy文件,方便在主程序中读取1
2
3
4
5
6
7
8# create_matrix.py
import numpy as np
matrix = np.random.randint(20, 200 + 1, (100, 100))
matrix = np.triu(matrix, k=1)
matrix = matrix + matrix.T
np.save("distance_matrix.npy", matrix)在
TSP_GA.py脚本中,编写类TSP_GA和其内部类Individual的初始化函数。在初始化时,需从npy文件中导入距离矩阵,并生成第一代种群,每个个体的DNA都是随机生成的。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37# TSP_GA.py
import numpy as np
import matplotlib.pyplot as plt
import os
import pickle
class TSP_GA():
def __init__(self, city_num, carrying_capacity, survival_rate, crossover_rate, mutation_rate):
self.city_num = city_num
self.carrying_capacity = carrying_capacity
self.survival_rate = survival_rate
self.crossover_rate = crossover_rate
self.mutation_rate = mutation_rate
self.average_distance_list = []
self.generation_counter = 1 # 代(generation)数
self.not_updated_counter = 0 # 未更新最优解的代(generation)数
self.load()
self.generate_first_generation()
class Individual():
def __init__(self, outer):
self.outer = outer
self.dna = np.random.permutation(self.outer.city_num)# dna为整数0~citynum-1的随机排序列表,代表了到达的城市顺序
self.update_total_distance()
def update_total_distance(self):
self.total_distance = np.sum([self.outer.distance_matrix[self.dna[i]][self.dna[(i+1)%(self.outer.city_num)]] for i in range(self.outer.city_num)])
def load(self):
self.distance_matrix = np.load("distance_matrix.npy")
def generate_first_generation(self):
self.individual_list = [self.Individual(self) for _ in range(self.carrying_capacity)]
self.individual_num = self.carrying_capacity
self.get_fitness_rates()为
TSP_GA类编写计算各个体适应度和从种群中选择存活个体的方法。对于后者,这里采取如下方法:有放回地从种群中选取个体加入存活个体集合,直到达到集合元素数量上限,即环境容纳量。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18# TSP_GA.py
class TSP_GA():
# ...
def get_fitness_rates(self):
self.fitness_list = [1 / self.individual_list[i].total_distance for i in range(self.individual_num)]
fitness_sum = np.sum(self.fitness_list)
self.fitness_rates = self.fitness_list / fitness_sum
if self.generation_counter == 1:
self.init_optimal_solution()
self.evaluation() # 每次计算完个体适应率后进行评估,更新最优解
def select(self):
selected_indeces = np.random.choice(np.arange(self.individual_num), size=self.carrying_capacity, replace=True, p=self.fitness_rates)
self.individual_list = [self.individual_list[i] for i in selected_indeces]
self.individual_num = len(self.individual_list)为
TSP_GA类编写交叉和变异的方法。1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41# TSP_GA.py
class TSP_GA():
# ...
def cross(self, father: Individual, mother: Individual):
# 使用PMX(部分映射交叉)方法来实现交叉
start_index = np.random.randint(0, self.city_num)
end_index = np.random.randint(start_index, self.city_num)
child = self.Individual(self)
child.dna = np.full(self.city_num, -1)
child.dna[start_index : end_index] = father.dna[start_index : end_index]
for i in range(self.city_num):
if child.dna[i] >= 0:
continue
for j in range(self.city_num):
if mother.dna[j] in child.dna:
continue
child.dna[i] = mother.dna[j]
break
child = self.mutate(child) # 子代概率变异
child.update_total_distance()
return child
def mutate(self, child: Individual):
# 概率发生变异
if np.random.rand() < self.mutation_rate:
mutation_index = np.random.randint(0, self.city_num)
mutation_value = np.random.randint(0, self.city_num)
return self.swap(child, mutation_index, mutation_value)
return child
def swap(self, individual: Individual, dna_index, swap_value):
# 将个体的dna_index位基因与值为swap_value的基因交换(目的是在改变dna的同时保证基因的唯一性,以保证能到达所有城市)
temp_value = individual.dna[dna_index]
temp_index = np.where(individual.dna == swap_value)
individual.dna[dna_index] = swap_value
individual.dna[temp_index] = temp_value
return individual为
TSP_GA类编写生成种群下一代的方法1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19# TSP_GA.py
class TSP_GA():
# ...
def generate_next_generation(self):
self.generation_counter += 1
self.select()
new_individual_list = []
for father in self.individual_list:
new_individual_list.append(father)
if np.random.rand() < self.crossover_rate:
mother = self.individual_list[np.random.randint(0, self.individual_num)]
child = self.cross(father, mother)
new_individual_list.append(child)
self.individual_list = new_individual_list
self.individual_num = len(self.individual_list)
self.get_fitness_rates()为
TSP_GA类编写寻找最优解的方法1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18# TSP_GA.py
class TSP_GA():
# ...
def init_optimal_solution(self):
self.optimal_solution = self.Individual(self)
self.optimal_solution = self.individual_list[np.argmax(self.fitness_list)]
def evaluation(self):
# 计算这一代的平均路程
self.average_distance_list.append(np.mean([self.individual_list[i].total_distance for i in range(self.individual_num)]))
# 检查是否更新最优解
temp_solution = self.individual_list[np.argmax(self.fitness_list)]
if temp_solution.total_distance < self.optimal_solution.total_distance:
self.optimal_solution = temp_solution
self.not_updated_counter = 0
self.not_updated_counter += 1编写主函数
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50if __name__ == '__main__':
city_num = 20 # 城市数目
carrying_capacity = 100 # 环境容纳量
survival_rate = 0.7 # 存活率
crossover_rate = 0.8 # 交叉率(平均每个个体诞下后代的个数)
mutation_rate = 1e-2 # 突变率
end_conditions = {
"generation_counter": 5000,
"not_updated_counter": 3000,
}
end_condition = "not_updated_counter"
# 运行算法
sol = TSP_GA(city_num=city_num, carrying_capacity=carrying_capacity, survival_rate= survival_rate, crossover_rate= crossover_rate, mutation_rate=mutation_rate)
if os.path.exists('result_counter.pkl'):
with open('result_counter.pkl', 'rb') as f:
result_counter = pickle.load(f)
else:
result_counter = 0
while getattr(sol, end_condition) < end_conditions[end_condition]:
if sol.generation_counter % 200 == 0:
print(f"generation {sol.generation_counter}: {sol.optimal_solution.total_distance}")
sol.generate_next_generation()
# 保存结果
x_values = np.linspace(0, len(sol.average_distance_list)-1, 100, dtype=int)
y_values = np.array(sol.average_distance_list)[x_values]
plt.xlabel("generation")
plt.ylabel("average_distance_of_each_generation")
plt.plot(x_values, y_values)
current_dir = os.path.dirname(os.path.abspath(__file__))
txt_path = os.path.join(current_dir, "./results.txt")
with open(txt_path, "a") as file:
result_counter += 1
file.write("result_" + str(result_counter) + ":\n")
file.write("city_num: " + str(city_num) + "\n")
file.write("carrying_capacity: " + str(carrying_capacity) + "\n")
file.write("crossover_rate: " + str(crossover_rate) + "\n")
file.write("mutation_rate: " + str(mutation_rate) + "\n")
file.write("end_condition: " + end_condition + "<" + str(end_conditions[end_condition]) + "\n")
file.write("Solution: " + str(sol.optimal_solution.dna) + "\n" + "Distance: " + str(sol.optimal_solution.total_distance) + "\n\n")
png_path = os.path.join(current_dir, f"./result_figures/result_{result_counter}")
plt.savefig(png_path)
with open('result_counter.pkl', 'wb') as f:
pickle.dump(result_counter, f)
结果
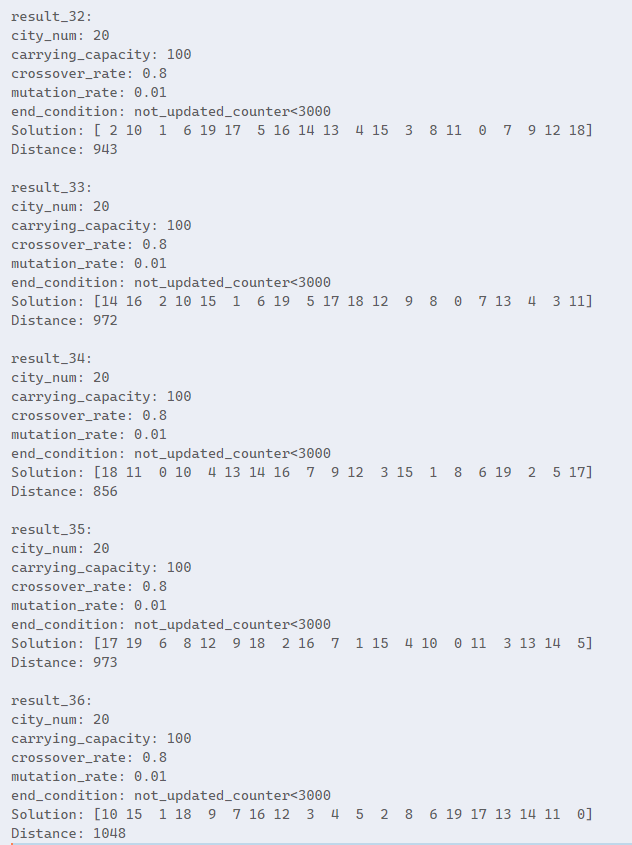
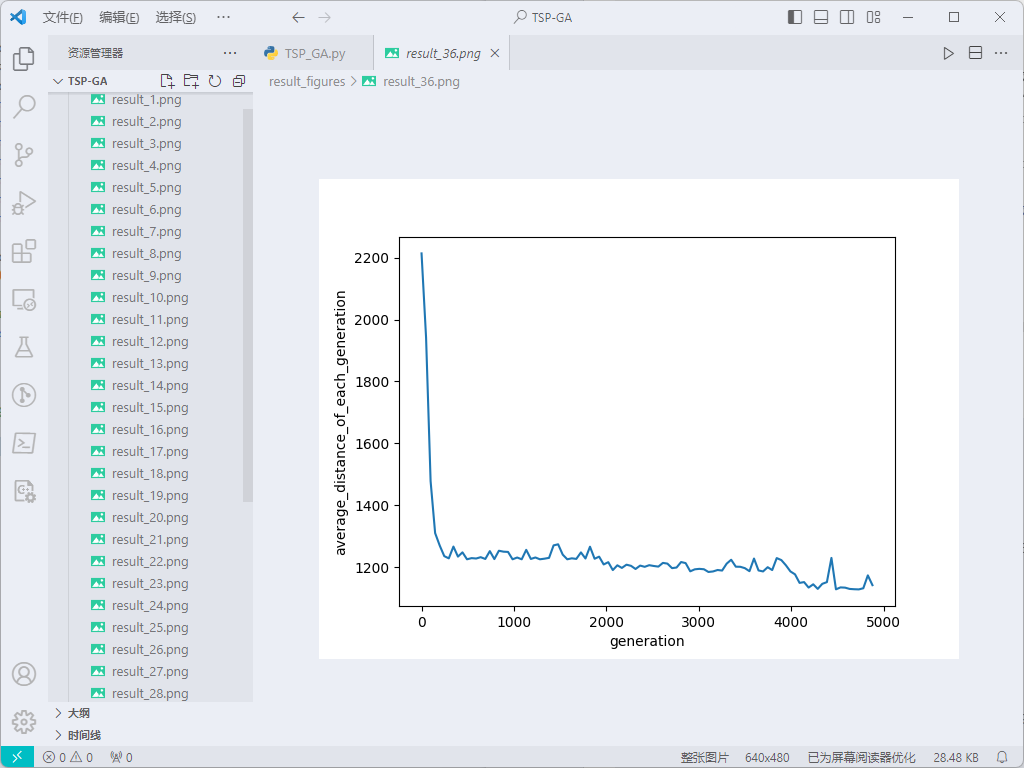
总结
- 尝试多次运行后,发现得到的结果并不稳定,且其优劣程度差异较大,问题可能出现在适应度的计算、选择的策略、交叉的方法等。
- 由于本人忙于摸鱼,在本次实践中有很多可以深入思考和优化的地方还未仔细斟酌,若有任何想法或问题,欢迎联系!